 Bilde: Skanning av en del av nervøst vev. Bioscience Image Library, Fayette Reynolds.
Bilde: Skanning av en del av nervøst vev. Bioscience Image Library, Fayette Reynolds.
I løpet av det siste året av graden min i datavisjon, skrev jeg en artikkel med tittelen “Know Your Neighbour”. I den fremhevet jeg hvordan teknikker innen maskinlæring kan brukes til å forbedre effektiviteten av det som kalles “3-dimensjonal neuronsegmentering”, altså prosessen med å identifisere nevroner i 3D-rom. Både jeg og veilederen min var veldig fornøyde med resultatene, så jeg bestemte meg for å skrive denne bloggen, som forklarer hva det er, hvordan det fungerer og hvilke implikasjoner det kan ha for fremtiden til nevrovitenskap.
I november 2022 publiserte forskere ved Cambridge University og Johns Hopkins University en tidlig versjon av artikkelen “The connectome of an insect brain”. Det var kulminasjonen av mange års arbeid og viste den første digitale rekonstruksjonen av den fulle hjernekoblingen (connectome) til en kompleks hjerne. Forskerne kartla møysommelig hver eneste nevron og hver eneste synapse hos en bananflue fra et datasett med 4841 elektronmikroskopi (EM) bilder.
Det var en enorm innsats og et banebrytende sprang for nevroforskere og biologer, som endelig hadde en komplett digital representasjon av en dyrehjerne. Ved å lage en algoritme som sporer signaler over hjernen, fikk forskerne innsikt i hvordan informasjon beveger seg mellom nevroner og hvilke deler av hjernen som er ansvarlige for hvilke sanser. For eksempel oppdaget forskerne at de fleste nevroner har mange roller, og er ansvarlige for mange forskjellige operasjoner samtidig. Med en bachelorgrad i kognitiv vitenskap, undret jeg meg umiddelbart over hvilken innvirkning en slik nevronal hjernekoblingskartlegging ville ha på forskningen på menneskelige hjerner. Kanskje det kunne tillate oss å få en bedre forståelse av kognitive sykdommer eller genetisk skadede nevrale baner.
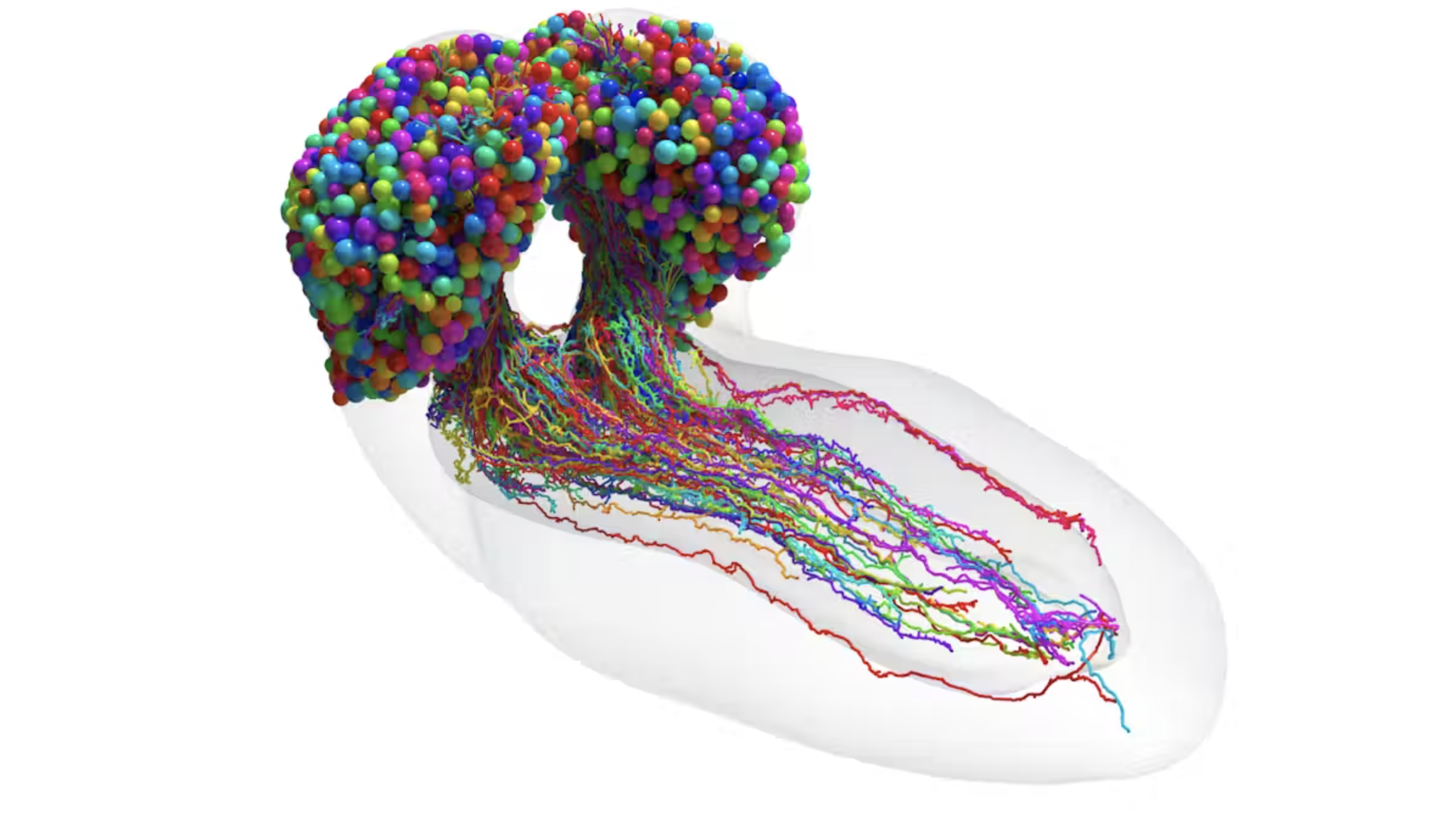 Bilde: Forskningsartikkel i Science
Bilde: Forskningsartikkel i Science
Min teori var at databehandling kunne brukes til å automatisere neuronsegmentering på EM-bilder fra menneskelige hjerner mer effektivt enn menneskelig annotering. Dette er selvsagt ikke en ny idé, men kanskje er det ikke helt åpenbart hvorfor det ikke har blitt gjort ennå. For det første hadde bananfluen som ble kartlagt bare 3016 nevroner og 548 000 synapser. Menneskehjernen, derimot, anslås å inneholde 86 milliarder nevroner. Hvor mange synapser har vi da? Over 100 billioner. For å sette det tallet i perspektiv, er det omtrent 1000 ganger så mange synapser i hjernen vår som det er stjerner i Melkeveien. Google utførte nylig den største hjerneskanningen noensinne på et 1 kubikkmillimeter stort område av hjernen. Rekonstruksjonen krevde mer enn 5000 vevsskiver som tok over ett år å produsere. Hva var størrelsen på det resulterende datasettet? 1,4 petabyte, tilsvarende 14 000 4K-filmer. Ekstrapolert videre, hvor mye lagringsplass ville vi trenge for å gjennomføre en komplett hjerneskanning? 1,6 zettabyte, med en kostnad på 50 milliarder amerikanske dollar og et areal på 140 mål, noe som ville gjort det til det største datasenteret på planeten – bare for ett bilde.
Så hvordan behandler vi så mange bilder? Min tilnærming er å unngå bruken av 3-dimensjonale bilder da de krever for mye datakraft. I stedet teoretiserte jeg at det ville være mye raskere – og viktigst av alt, like nøyaktig – å behandle hver skive separat og deretter projisere det i 3D-rom. Men hvordan kan programmet vite at en nevron i én skive er en fortsettelse av nevronen fra forrige skive? Her kommer hoveddelen av mitt akademiske bidrag inn. Jeg antok at vi kunne lære opp modellen til å vite hvilke deler av en skive som er koblet sammen ved å bruke en teknikk som heter “transfer learning”, eller “overført lærdom”. Transfer learning er en populær teknikk innen maskinlæring der kunnskap fra ett trinn overføres til neste trinn. Et vanlig brukstilfelle er når du bygger en modell som kan klassifisere nye klasser med lite eller ingen data, for eksempel når du trener en datavisjonsmodell til å gjenkjenne en sebra fra data om en hest.
 Et eksempel på hvordan en skive i datasettet mitt så ut.
Et eksempel på hvordan en skive i datasettet mitt så ut.
For å bevise hypotesen min testet jeg programmet på et stort datasett med 3D-nevronbilder ved hjelp av min prosesseringsteknikk. Etter litt forhåndsbehandling ble hvert 3D-bilde segmentert, og en resulterende liste over nevroner ble returnert. Suksess! Jeg prøvde også å kjøre en 3D-segmenteringsalgoritme på det samme datasettet. Resultatene? Programmet mitt var nesten dobbelt så raskt som 3D-algoritmen!
Ser vi fremover, er potensielle anvendelser av denne teknikken enorme. Ved å redusere tiden og de datamessige ressursene som kreves for nevronsegmentering, åpner vi døren for mer omfattende og detaljerte studier av menneskehjernen. Dette fremskrittet kan akselerere forskningen på nevrologiske lidelser, kognitive sykdommer og utviklingen av mer sofistikerte hjerne-datamaskin-grensesnitt. Jeg tror ikke denne teknikken vil bli brukt på et fullstendig datasett med det første, da det er mange flaskehalser som må løses før en menneskelig hjernekoblingskartlegging er oppnåelig. Når det er sagt, tror jeg nye fremskritt innen KI er avgjørende for vår forståelse av sammenkoblingen av menneskelige hjerner. Jeg ser frem til å se hvor vitenskapen tar oss.
– Sigurd